Labordiagnostische Begleituntersuchungen im Rahmen der Ausbruchsaufklärung

Ein zentrales Element in der Erkennung und Bewältigung von Ausbrüchen ist die Identifizierung und genaue Charakterisierung der ursächlichen Erreger. Um eine möglichst zielgerichtete Probenentnahme und anschließende Laboranalyse planen und durchführen zu können, ist das Vorliegen aller bereits vorhandenen Daten und Informationen notwendig. Wichtig sind in diesem Zusammenhang z.B. Angaben zum Krankheitsverlauf, zur Krankheitsdauer, zu der Anzahl der betroffenen Personen, zu den Ergebnissen von bereits vorliegenden Laboruntersuchungen sowie von epidemiologischen Untersuchungen, um Hinweise auf den potenziellen Erreger, den vermutlichen Infektionszeitpunkt und den Infektionsort zu erhalten. Labordiagnostische Untersuchungen können auch erste Hinweise auf einen Ausbruch liefern, wenn sich bei der genauen molekularbiologischen Charakterisierung unterschiedlicher Patientenproben zeigt, dass eine bestimmte Erregervariante gehäuft vorkommt.
Labordiagnostische Leistungen in diesem Zusammenhang werden u.a. von erregerspezifischen Nationalen Referenzzentren und Konsiliarlaboren vorgenommen (Link siehe unten). Insgesamt gibt es einschließlich der am RKI ansässigen Referenzlabore deutschlandweit 21 Nationale Referenzzentren und 39 Konsiliarlabore, die den Öffentlichen Gesundheitsdienst und das RKI bei ergänzenden Untersuchungen im Rahmen von Ausbruchsuntersuchungen unterstützen. Diese Labore stellen wesentliche Elemente des Infektionsschutzes dar und ergänzen durch die erregerspezifische Fachkompetenz die infektionsepidemiologische Surveillance (Überwachung). Die Untersuchung von Lebensmittelproben obliegt dem Bundesinstitut für Risikobewertung (BfR) und den Landesbehörden der Lebensmittelüberwachung. Im Rahmen von Ausbruchsuntersuchungen arbeitet das RKI eng mit diesen Behörden zusammen.
Ziel der Untersuchungen ist zunächst das Identifizieren des Erregers. Treten neue Erreger auf, müssen die Testverfahren unter Umständen erst entwickelt oder angepasst werden. Durch die Untersuchung geeigneter Patientenproben mit etablierten Tests kann festgestellt werden, ob ein Patient sich mit dem Erreger angesteckt hat. Dabei ist auch das Zeitfenster für den Nachweis zu beachten. Handelt es sich bei einer Ausbruchsursache um einen neuen Erreger, wie zum Beispiel das SARS-Coronavirus, ist bei einem entsprechenden labordiagnostischen Nachweis schnell klar, dass der Patient zum Ausbruchsgeschehen zu zählen ist. Insbesondere bei lebensmittelbedingten Krankheitsausbrüchen wird die Aufklärung von Infektketten durch zusätzliche molekularbiologische Methoden zur Feintypisierung, wie z.B. durch Sequenzierung, ermöglicht. So können über den Nachweis der klonalen Identität von Erregern aus einer vermuteten Infektionsquelle und dem Patienten Aussagen zur Infektkette im konkreten Fall gemacht werden. Bei Erregern wie beispielsweise dem 2018 aufgetretenen Listeria monocytogenes-Klon „Sigma1" war eine molekularbiologische Charakterisierung erforderlich, um zu differenzieren, ob Patienten sich mit dem Ausbruchsstamm oder mit anderen zirkulierenden L. monocytogenes-Bakterien angesteckt hatten.
Methoden
Im Labor werden unterschiedliche Methoden zum Nachweis und zur Charakterisierung der Erreger verwendet, bei Ausbruchsgeschehen stehen meist die folgenden im Mittelpunkt:
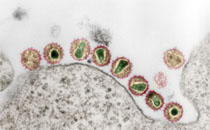
Mikroskopische Verfahren: Die elektronenmikroskopische Untersuchung von Probenmaterial auf Krankheitserreger ist eine schnelle, im optimalen Fall innerhalb einer Stunde durchführbare Methode, die Orientierung in Bezug auf eine erste Diagnose bietet. Sie erfasst alle Partikel einer Probe (offener Blick), inklusive der kleinsten Viren, und kann damit sowohl bisher bekannte, als auch unbekannte Erreger (bis zur Auflösungsgrenze von etwa einem Nanometer) entdecken. Da die Erregerbestimmung relativ unspezifisch ist und für den Nachweis höhere Konzentrationen der Erreger notwendig sind, wird die Mikroskopie nur in bestimmten Fällen eingesetzt. Die mikroskopische Untersuchung ergibt im Idealfall Hinweise für eine zielgerichtete und spezifische weitere Diagnostik, den Ausschluss von vermuteten Erregern oder die unabhängige Kontrolle sowie Bestätigung von Diagnosen, die mit anderen Methoden erzielt wurden.
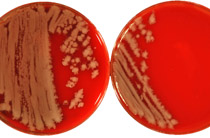
Anzucht: Erreger müssen oft erst isoliert und vermehrt werden, damit genügend Material in Reinform für weiterführende Analysen, wie z.B. der Resistenztestung oder der molekularbiologischen Charakterisierung, zur Verfügung steht. Eine solche Anzucht kann mehrere Stunden oder Tage, in seltenen Fällen auch Wochen dauern. Viren müssen auf Zellkulturen angezogen werden, da sie sich nicht selbstständig vermehren können; für Bakterien, Pilze oder Parasiten sind entsprechende Kulturmedien ausreichend.
Resistenztests: Im Labor wird überprüft, ob ein Erreger gegen Antiinfektiva empfindlich ist oder Resistenzen aufweist, die eine Therapie erschweren. Das ist für die behandelnden Ärztinnen und Ärzte eine wichtige Information. Resistenztests sind in der Regel erst nach Erregeranzucht möglich.
Molekularbiologische Charakterisierung
- PFGE: Die sogenannte Pulsfeldgelelektrophorese (PFGE) wird bei bakteriellen Erregern verwendet und dient der Typisierung der Isolate. Dabei wird das Erbgut durch molekulare „Scheren“ in kleine Stücke geschnitten und in einem elektrischen Feld (auf einem Gel) aufgetrennt. Es entstehen Bandenmuster, die für einzelne Bakterienstämme charakteristisch sind, sodass ein Ausbruchsstamm von anderen Stämmen des gleichen Bakteriums unterschieden werden kann.
- Sequenzierung: Bei der Sequenzierung wird die Reihenfolge der Erbgutbausteine bestimmt. Es werden Erbgutabschnitte sequenziert, die für den Nachweis von z.B. Resistenzgenen und/oder zur Unterscheidung der Erregervariante von verwandten Varianten wichtig sind.
- Ganzgenomsequenzierung: Heutzutage stellt die Sequenzierung des kompletten Erbguts eines bakteriellen oder viralen Erregers die Methode der Wahl zur Bestimmung von Verwandtschaftsverhältnissen der Erreger zueinander und deren Abstammung (Phylogenie) dar. Die so erhaltenen Sequenzdaten können mit vorhandenen Referenzsequenzen durch die Identifizierung der Einzelnukleotidunterschiede (im Englischen Single Nucleotide Polymorphism, SNP) im Erbgut abgeglichen werden. Bei bakteriellen Erregern hat sich vor allem die Kerngenomanalyse durchgesetzt. Sie betrachtet, in Abhängigkeit vom Erreger, die Nukleotidsequenzen und deren Unterschiede von mehreren hundert kodierenden Regionen des Erbguts. Beide Verfahren zählen zu den hochauflösenden Typisierungsmethoden und bieten eine bessere Diskriminierungsfähigkeit als PFGE oder die Sequenzierung von nur einzelnen genetischen Abschnitten.
Serologische Untersuchungen: Durch den Nachweis von Antikörpern im Blut eines Patienten kann festgestellt werden, ob eine akute Infektion vorliegt (IgM-Nachweis), oder ob durch frühere Infektionen oder Impfungen einen Schutz gegen den Erreger besteht (IgG-Nachweis).
Der kulturelle labordiagnostische Nachweis von Erregern bei Erkrankten ist anzustreben. Zudem können die so gewonnen Erregerkulturen der molekularbiologischen Erregerüberwachung zugeführt und in den Kontext mit epidemiologischen Daten gebracht werden (Integrierte Genomische Surveillance). In einem Ausbruchsgeschehen ist es nicht immer möglich, Proben zu nehmen oder auf die ursprünglichen, an die Nationalen Referenz- bzw. Konsiliarlabore eingesandten Proben für eine weitergehende Erregerfeintypisierung zurückzugreifen. In Abhängigkeit von der Inkubationszeit, der Ausscheidungsdauer und der Erkennung einer Häufung muss eine vom Gesundheitsamt angeordnete Probennahme auch ein Erreger-spezifisches Zeitfenster treffen. Ansonsten kann der Erregernachweis falsch-negativ ausfallen.
Darüber hinaus ist das Erkennen eines epidemiologischen Zusammenhanges in großen, geografisch weiträumigen Krankheitsausbrüchen für die meisten Fälle oft nur anhand des Erregernachweises bzw. - bei relativ häufigen Krankheitserregern - der Feintypisierung der Erreger möglich. Im Falle der großen Ausbrüche mit den L. monocytogenes-Klonen Sigma1 (2014-19) und Ny9 (2020-21), EHEC O104 (2011), Noroviren (2012) und Masernviren (2013) gelang unter Beteiligung der entsprechenden Referenz- bzw. Konsiliarlabore der Nachweis der beteiligten Erreger bei einer Vielzahl von Betroffenen mittels molekularbiologischer und sequenzbasierter Verfahren, so dass der Zusammenhang der Erkrankungen eindeutig belegt werden konnte.
nach oben
Weitere Informationen
nach oben