Forschungsprojekte
Erhöhung der Aussagekraft und des Public Health Nutzens der Integrierten Genomischen Surveillance von HIV mittels Automatisierungen und Erweiterungen
Im Rahmen eines BMG-geförderten Projekts wird die IGS-HIV (ehemals MolSurv-HIV) weiterentwickelt, um die deutsche HIV-Epidemie kontinuierlich bewerten und eine evidenzbasierte Gestaltung von Public Health-Maßnahmen in den Bereichen Prävention, Diagnostik und Therapie zu ermöglichen. Hierfür sind bis Ende 2025 verschiedene labortechnische und bioinformatische Automatisierungen und Erweiterungen geplant (s. Abbildung 1, grün markiert). Das Vorhaben schließt die molekulare Vollerfassung aller HIV-Neudiagnosen ein, wofür auch die Übermittlung vorliegender HIV-Sequenzen aus der genotypischen Resistenzbestimmung in Diagnostiklaboren zusammen mit der Meldung via DEMIS geprüft wird. Der Laborprozess im HIV-Studienlabor soll automatisiert und die Sequenzierung auf das HIV-Vollgenom erweitert werden. Für die Auswertung der NGS-Daten und die nachfolgenden Analysen mit den HIV-Sequenzen sollen automatisierte bioinformatische Pipelines aufgesetzt werden. Dies umfasst auch die Evaluation einer bioinformatischen Methode zur Bestimmung der Rezenz bzw. Infektionsdauer in der InzSurv-HIV über die HIV-NGS-Sequenz. Quartalsweise sollen die Ergebnisse zur Primärresistenz, zur Diversität, zu Transmissionsclustern und Ausbrüchen sowie zur Rezenz automatisiert ausgewertet und über ein Dashboard der Öffentlichkeit präsentiert werden.
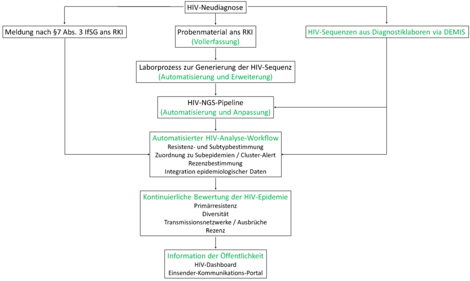
 Abbildung 1: Geplanter Workflow der IGS-HIV. Entsprechende Erweiterungen sind grün markiert. Quelle: © RKI
Abbildung 1: Geplanter Workflow der IGS-HIV. Entsprechende Erweiterungen sind grün markiert. Quelle: © RKI
Evaluierung eines automatisierten AmpliSeq-Verfahrens zur HIV-Sequenzgenerierung bei steigenden HIV-Neudiagnosen aufgrund des Ukraine-Kriegs
Im Rahmen der IGS-HIV werden derzeit ca. 60% aller HIV-Neudiagnosen im HIV-Studienlabor molekularbiologisch untersucht. Der aktuelle Laborprozess basiert auf der Amplifizierung und Sequenzierung (NGS) einzelner HIV-Genomabschnitte ausgehend von Virus-RNA. Der Laborprozess ist jedoch nur in Teilen automatisiert und die arbeits- und zeitintensive Bearbeitung der Proben erlaubt weder eine Steigerung des Probendurchsatzes noch eine Ausweitung auf das HIV-Vollgenom. Daher wird für die IGS-HIV eine Automatisierung des Laborprozesses angestrebt, die eine Vollerfassung der jährlich etwa 3.000-3.500 HIV-Neudiagnosen in Deutschland ermöglicht. Vor dem Hintergrund der ukrainischen Flüchtlingsbewegung gewinnt dieses Ziel nochmals an Bedeutung, um die Auswirkungen der Ukraine-Kriegs auf die HIV-Epidemie in Deutschland möglichst umfassend überwachen und zielgerichtete Präventions- und Public Health-Maßnahmen einleiten zu können.
Ziel des Projektes ist es, einen automatisierten Laborprozess aufzubauen, der bei steigendem Probenaufkommen und gleichbleibend guten Sequenz-Ergebnissen zu einer Reduzierung der Arbeitslast und somit einer Steigerung der Wirtschaftlichkeit führt. Hierzu werden zwei Verfahren zur Amplikon-Sequenzierung (AmpliSeq) der Firmen Thermo Fisher Scientific und Illumina verglichen. Des Weiteren wird ein Ansatz zum Viral Capturing mit Illumina Sequenzierung evaluiert.
Detektion von Ursachen für den Anstieg von HIV-Infektionen in injizierenden Drogengebrauchenden in Deutschland
In Deutschland kommt es immer wieder zu Ausbrüchen oder schnell wachsenden Subepidemien, die räumlich eng begrenzt sind, jedoch in der generellen Surveillance aufgrund ihrer relativ geringen Fallzahlen nicht gesehen werden können. Diese Cluster und Subepidemien fallen oftmals erst in späteren Detail-Analysen auf, wodurch mögliche Präventionsmaßnahmen verzögert erfolgen oder gar nicht mehr anwendbar sind. Dazu gehören beispielsweise Cluster injizierender Drogengebrauchender (people who inject drugs, PWID), in denen sich das Virus über einen kurzen Zeitraum lokal stark ausbreitet. Das frühzeitige, automatisierte Erkennen dieser schnell wachsender Infektionscluster wäre enorm hilfreich, um beizeiten in den entsprechenden Regionen präventiv aktiv werden zu können und Transmissionswege frühzeitig zu unterbrechen.
Ziel des Projektes ist es, neue HIV-Sequenzen schnell-wachsender Cluster in den jeweiligen Transmissionsgruppen schnell zu detektieren und entsprechend zuzuordnen. Aufgrund der großen Datenmenge erfolgt die Detektion automatisiert basierend auf der Software HIV-TRACE. Nach vorheriger automatischer Vorsortierung entsprechend der Subtypen der zu untersuchenden Sequenzen, sollen diese an HIV-TRACE übergeben werden. Es soll eine automatisierte Clusterzuordnungs-Pipeline etabliert werden, in der die verschiedenen Subepidemien von HIV als Referenzdatensätze hinterlegt sind. Testdatensätze sollen verwendet werden, um die richtige Zuordnung von Sequenzen zu verifizieren. Anschließend sollen Sequenzen aus dem Jahr 2021 und 2022 auf Zugehörigkeit zu bekannten PWID-Clustern untersucht werden, um zu prüfen, inwieweit Ausbruchsgeschehen für den Anstieg der Meldungen in dieser Risikogruppe verantwortlich sind.
Entwicklung einer phylodynamischen Methode für die Analyse von HIV-Rekombinanten und Charakterisierung von HIV-Rekombinationsereignissen in der deutschen Epidemie
Rekombination bei Multi- oder Superinfektionen ist eine wesentliche Eigenschaft von HIV und hat einen bedeutenden Einfluss auf die Evolution der HIV-Epidemie. Aus individuellen URF (unique recombinant form) können durch erfolgreiche Weiterübertragung epidemisch relevante CRF (circulating recombinant forms) entstehen. Durch weitere Rekombination von CRF bilden sich komplexe rekombinante Formen. Molecular Clock Phylogenetic Inference-Methoden erlauben keine Analyse von Transmissionsnetzwerken und Reproduktionsraten mit rekombinanten HIV. In einem Doktorandenprojekt mit dem „Zentrum für Künstliche Intelligenz in der Public Health-Forschung“ (ZKI-PH 2, Dr. Denise Kühnert) soll daher eine Bayes Methode aufgesetzt werden, die es ermöglicht das Übertragungspotential und die Evolution von HIV-Rekombinanten zu bewerten.
Auswirkung der HIV-bedingten Schädigung des Immunsystems auf die HIV/SARS-CoV-2-Koinfektion und SARS-CoV-2-Vakzinierung (HIVCOV)
Im Vergleich mit der Allgemeinbevölkerung zeigen Menschen mit HIV schwerere COVID-19-Verläufe sowie signifikant geringere Konzentrationen an anti-SARS-CoV-2 IgG und neutralisierenden Antikörpern. Im HIVCOV-Projekt wurde daher die die Vulnerabilität von Menschen mit HIV gegenüber SARS-CoV-2-Infektionen und der Wirksamkeit von anti-SARS-CoV-2-Vakzinen untersucht, um einen Beitrag zur Erarbeitung von z.B. Vakzinierungsstrategien für Menschen mit HIV zu leisten.
Zunächst wurden in der HIV-1 Serokonverter-Kohorte die SARS-CoV-2 anti-S sowie anti-N positive Fälle im Zeitraum Q1/2020-Q3/2021 identifiziert. Nachfolgend wurden die betreffenden Kohortenmitglieder im Q4/2021 eingeladen am HIVCOV-Projekt teilzunehmen. Anhand von im Jahr 2022 quartalsweise eingesammelten Blutproben werden derzeit die Verläufe der humoralen (anti-S IgG, neutralisierende Antikörper) und zellulären Immunantworten (T-Zellaktivierung) nach einer SARS-CoV-2-Infektion bei den HIVCOV-Teilnehmenden untersucht. Gleichzeitig erfolgte ab dem Q4/2021 eine klinisch-epidemiologische Erhebung zu SARS-CoV-2 Impfungen und Infektionen. Insgesamt konnten von 251 bzw. 1042 Teilnehmenden der HIV-1 Serokonverterstudie Proben bzw. Daten für das HIVCOV-Projekt gewonnen werden.
nach oben