Virologische Analysen im Nationalen Referenzzentrum für Influenzaviren Saison 2024/25
Stand: 16.04.2025
Genetische Charakterisierung der Influenzaviren
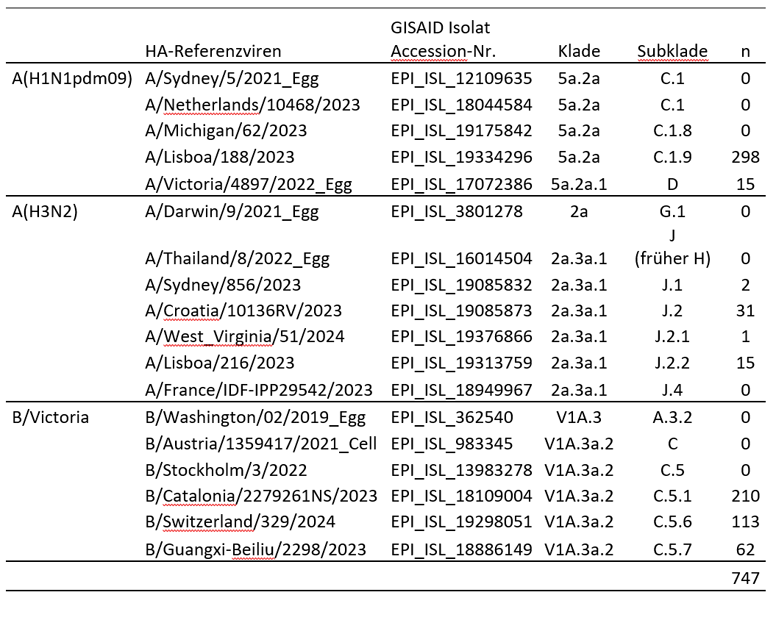
Aus Proben des ARE-Praxis-Sentinels (n = 656 blau), des SARI-Krankenhaus-Sentinels (n = 57, rot) sowie aus Proben von anderen Einsendungen (n = 34, lila) wurden 747 Influenzaviren der Saison 2024/25 sequenziert und das Hämagglutinin (HA) genetisch analysiert (Abb. 1-3). Unter den sequenzierten Influenzaviren gehörten 313 zum Subtyp A(H1N1)pdm09, 49 zum Subtyp A(H3N2) und 385 Viren zur B/Victoria-Linie. Entsprechend ECDC/WHO-Richtlinien wurden die Viren zu Referenzviren und entsprechenden Kladen und Subkladen zugeordnet (Tabelle 1).
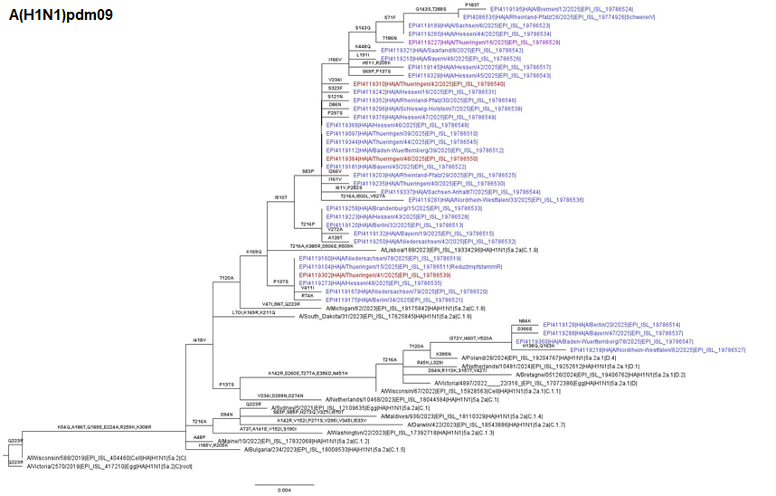
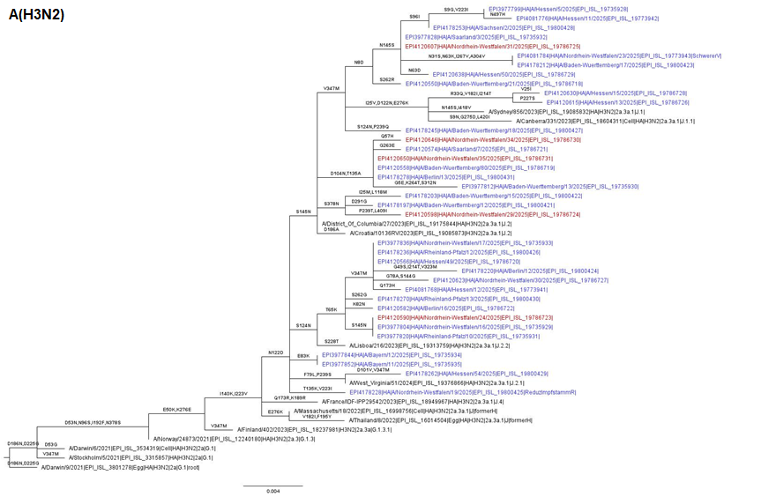
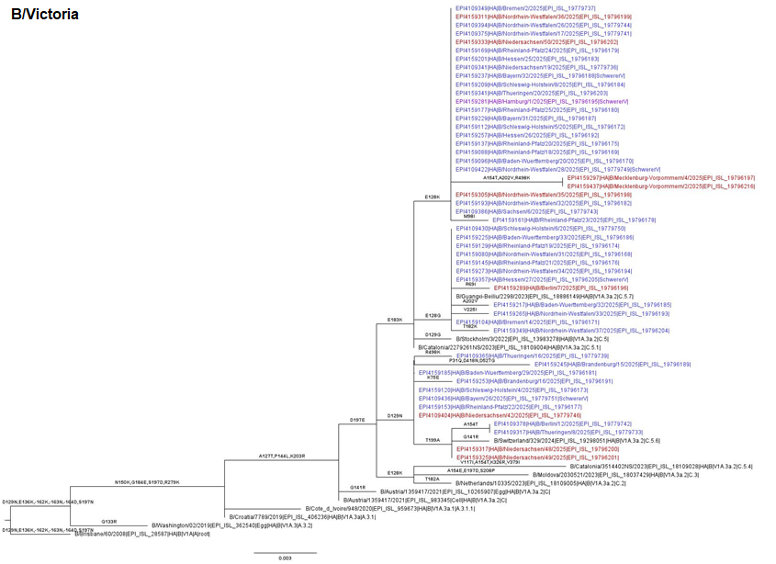
Abb. 1-3: Phylogenetische Analyse des Hämagglutinin (HA)-Gens von repräsentativen A(H1N1)pdm09-, A(H3N2)- und B/Victoria-Viren aus 2024/25. Die Proben wurden in den Kalenderwochen 04/2025 – 08/2025 entnommen. Die Virusgenome wurden aus Patientenproben vom ARE-Praxis-Sentinel (blau), SARI-Krankenhaus-Sentinel (rot) und von anderen Einsendungen (lila) gewonnen, über Next Generation Sequencing (NGS) sequenziert und die Vollgenome unter www.gisaid.org hinterlegt. Influenzaviren von Patientinnen und Patienten mit einem schweren bis fatalen Verlauf (SchwererV), z. B. aufgrund einer Pneumonie und Influenzaviren von geimpften Patientinnen und Patienten (Impfdurchbruch/ImpfD) sind bevorzugt abgebildet. Weiterhin sind Influenza A-Viren mit reduzierter Reaktivität gegenüber dem Impfstoffstamm (ReduzImpfstammR) dargestellt. Darüber hinaus sind ECDC/WHO-Referenzviren und Referenzviren, die für die genetische Zuordnung der zirkulierenden Viren bestimmt wurden, abgebildet. Die abgeleiteten Aminosäuresubstitutionen und Deletionen (-) im HA sind angegeben.
Methode phylogenetische Analyse: Mafft v. 7.520 (Accuracy-based Algorithmus: E-INS-i) für die Erstellung eines Multiplen Sequenz Alignments; Iqtree v. 2.2.2.7 (ML Methode) mit automatischer Selektion des Substitutionsmodels (FLU+I für H0N0, H3N2 und FLU+G4 für H1N1) und 1000 Bootstrap Wiederholungen für die Erstellung der Phylogenie; TreeTime v. 0.11.1 für die Rekonstruktion anzestraler Sequenzen und Annotation der Zweige mit Mutationen.
Tabelle 1: HA-Analyse und Zuordnung der zirkulierenden Influenzaviren in 2024/25 (Probeneingang ab KW40/2024) zu Referenzviren und Kladen (ECDC/WHO)
Antigenetische Charakterisierung der Influenzaviren
In der Saison 2024/2025 wurden bisher 280 Influenza A(H1N1)pdm09-Viren, 125 Influenza A(H3N2)-Viren und 237 Influenza B-Viren der Victoria-Linie in Zellkultur isoliert. 147 A(H1N1)pdm09-Virusisolate, 38 A(H3N2)-Isolate und 169 B/Victoria-Virusisolate wurden von anderen Einrichtungen eingesandt und in die antigene Charakterisierung einbezogen. Insgesamt wurden 996 Influenzavirusisolate untersucht. Die A(H1N1)pdm09-Viren reagierten gut mit dem entsprechenden Referenzserum (Impfstoffvirus A/Victoria/4897/2022; Abb. 4), jedoch gab es im März 2025 häufiger Reagenten außerhalb des Bereiches optimaler Passgenauigkeit. Alle isolierten A(H3N2)-Viren wurden vom gegen den Impfstamm gerichteten Referenzserum (Impfstoffvirus A/Thailand/8/2022) im Hämagglutinationshemmtest sehr gut erkannt (Abb. 5). Die B/Victoria-Viren wurden vom Impfstammantiserum (B/Austria/1359417/2021) ebenfalls detektiert, reagierten aber größtenteils schwächer als das Impfstammvirus (Abb. 6). Das bedeutet, dass die zirkulierenden B/Victoria Viren noch gut von gegen den Impfstamm gerichteten Antikörpern erkannt werden, jedoch mit einer kürzeren Dauer der Immunität nach Impfung zu rechnen ist.
Diese Analysen dienen der Untersuchung der Passgenauigkeit der Impfstämme; sie erlauben keine vollständigen Aussagen zur Wirksamkeit der Impfstoffe, da hier noch andere Faktoren berücksichtigt werden müssen (Abstand zur letzten Impfung, Zahl vorangegangener Antigenkontakte, Expositionsdosis, Alter u.a.).

Abb. 4: Reaktivität isolierter A(H1N1)pdm09-Influenzaviren mit gegen den Impfstamm A/Victoria/4897/2022 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen das Impfstammvirus); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm.
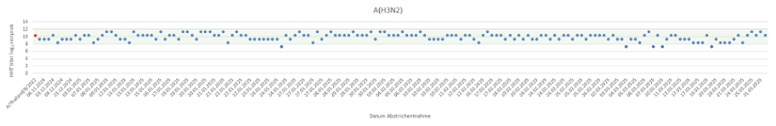
Abb. 5: Reaktivität isolierter A(H3N2)-Influenzaviren mit gegen den Impfstamm A/Thailand/8/2022 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen den Impfstamm); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm.

Abb. 6: Reaktivität isolierter B/Victoria-Influenzaviren mit gegen den Impfstamm B/Austria/1359417/2021 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen den Impfstamm); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm.
Resistenzanalyse der Influenzaviren
Von 327 untersuchten Influenzaviren zeigten sich 267 gegen die Neuraminidasehemmer Oseltamivir und Zanamivir empfindlich:
152 A(H1N1)pdm09
73 A(H3N2)
101 Influenza B-Victoria.
Ein A(H1N1)pdm09 Isolat zeigte aufgrund der NA-H275Y Substitution eine hohe Resistenz gegenüber Oseltamivir.
Mutationen, die mit einer Resistenz gegenüber dem Polymerasehemmer Baloxavir marboxil assoziiert sind, wurden in den 426 mit NGS untersuchten Influenzaviren nicht detektiert.
Immunitätslage vor der Saison
Es wurden jeweils 100 Seren der jeweiligen Altersgruppe im August 2024 mittels Hämagglutinationshemmtest gegen die Impfstoffviren der Saison 2024/2025 untersucht. Dabei zeigten sich hohe Seroprävalenzen gegenüber den Influenza A Viren, insbesondere bei der Altersgruppe der 5-15-Jährigen. Gegenüber den Influenzaviren der B/Victoria-Linie war die Immunität bei den jüngeren Altersgruppen gering (Abb. 7-9).
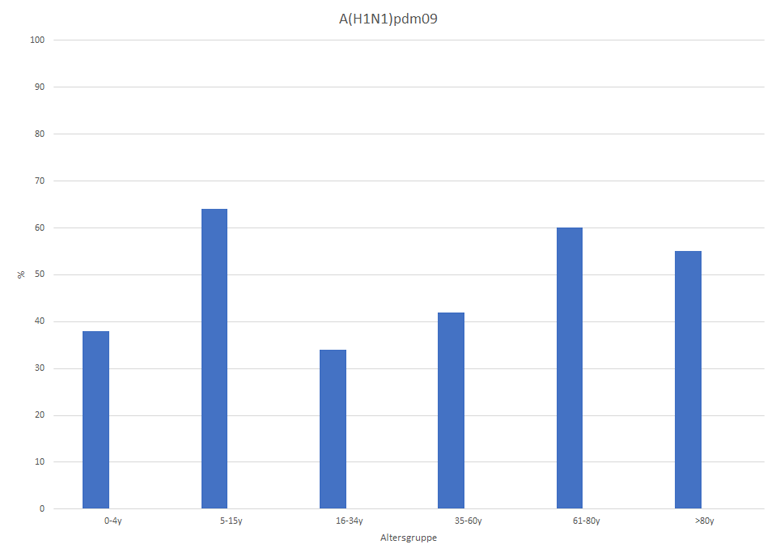
Abb. 7: Seroprävalenzen gegenüber A(H1N1)pdm09 (August 2024, Raum Berlin)
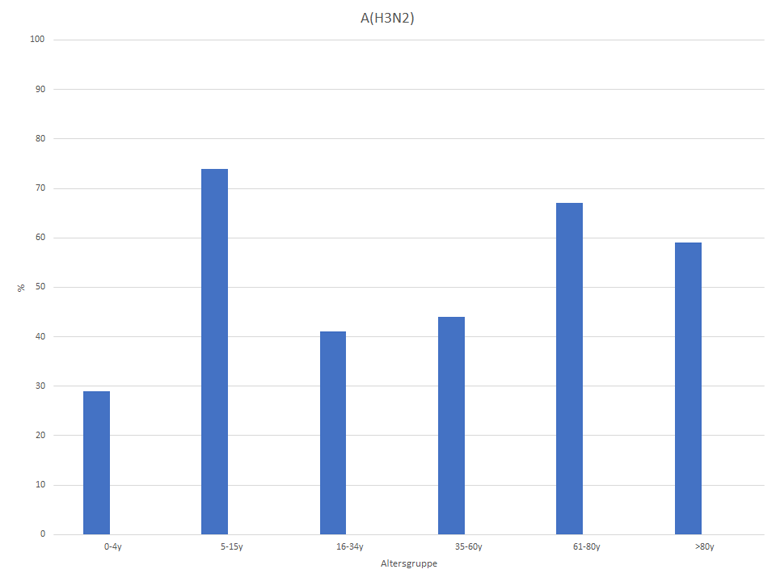
Abb. 8: Seroprävalenzen gegenüber A(H3N2) (August 2024, Raum Berlin)
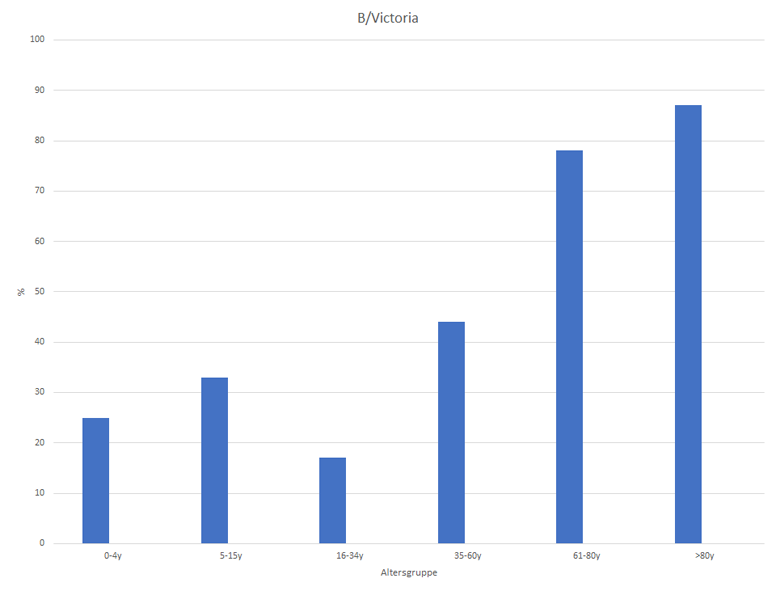
Abb. 9: Seroprävalenzen gegenüber B/Victoria (August 2024, Raum Berlin)
Zirkulation respiratorischer Viren
Die Grippewelle 2024/2025 begann in der ersten Kalenderwoche 2025. Sie wurde bis zur Kalenderwoche 5 2025 von A(H1N1)pdm09 Viren dominiert. Der Gipfel der A(H1N1)pdm09 Influenzaviruszirkulation war mit Positivenraten von 29% in der Kalenderwoche 4 2025 erreicht. Influenzaviren der B/Victoria-Linie zirkulieren parallel und hatten zeitversetzt zu den A(H1N1)pdm09 Viren ihren Peak in Kalenderwoche 7 2025 mit Positivenraten von 36%. A(H3N2) Viren werden seit der Kalenderwoche 47 2024 mit Positivenraten von 1-9% nachgewiesen. Derzeit kumulieren sich die Positivenraten für Influenzaviren auf 9% (Kalenderwoche 15 2025) und sind damit rückläufig (Kalenderwoche 13 2025: 17%) (Abb. 10 + Abb. 13). Das Ende der Grippewelle deutet sich an. Mit Beginn der Grippewelle waren die Nachweise von Humanen Rhinoviren zurückgegangen; mit der Kalenderwoche 15 2025 haben sie wieder das übliche Niveau von >15-20% erreicht (Abb. 11). Die Nachweise von Humanem Metapneumovirus (HMPV) liegen gegenwärtig (Kalenderwoche 15 2025) bei 8% der untersuchten Proben; Parainfluenzaviren (PIV) und Adenoviren (AdV) zirkulieren derzeit mit Positivenraten von 7 und 4%. Die Positivenraten für Respiratorisches Syncytialvirus (RSV) liegen gegenwärtig um 16%; die RSV-Welle hat mit der dritten Kalenderwoche 2025 begonnen, entfaltete sich aber nicht wie in den Vorjahren und erreichte erst in den Kalenderwochen 10 bis 15 mit 11-16% die bisher höchsten Positivenraten (Abb. 11). Die Zirkulation von Coronaviren war bei SARS-CoV-2 durch eine ähnliche Welle wie im Jahr zuvor charakterisiert, die Positivenraten gehen seit Mitte Dezember zurück und sind gegenwärtig auf einem sehr niedrigen Niveau. Bei den saisonalen Coronaviren zirkuliert vorwiegend OC43 (Abb. 12).
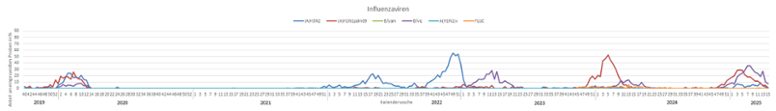
Abb. 10: Zirkulation von Influenzaviren von der letzten präpandemischen Saison bis Kalenderwoche (KW) 15 2025 (ARE-Praxis-Sentinel)
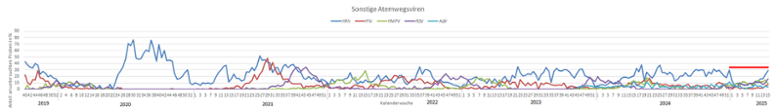
Abb. 11: Zirkulation von Rhinoviren (HRV), Parainfluenzaviren (PIV), Humanen Metapneumoviren (HMPV) und Respiratorischen Synzytialviren (RSV) von der letzten präpandemischen Saison bis KW15 2025, roter Balken: Periode geringer Nachweisraten von HRV
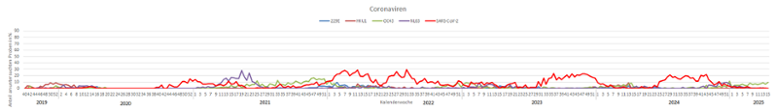
Abb. 12:Zirkulation von SARS-CoV-2 und endemischen Coronaviren (229E, HKU1, OC43, NL63) von der letzten präpandemischen Saison bis KW15 2025
Charakteristika der Grippewelle 2024/25
In Bezug auf die kumulativen Nachweisraten von Influenza A und B Viren war die Grippewelle 2024/25 die zweitstärkste nach 2017/18 (Abb. 13). Über sieben aufeinanderfolgende Wochen lagen die kumulativen Positivenraten über 50%. Die Grippewelle 2024/25 war durch eine weitgehend parallele Zirkulation von A(H1N1)pdm09 und B/Victoria Viren bei schwacher Zirkulation von A(H3N2) Viren gekennzeichnet, wobei der Gipfel der Viruszirkulation von A(H1N1)pdm09 dem von B/Victoria vorausging (Abb. 14). Die A(H1N1)pdm09-Positivenraten auf dem Peak der Viruszirkulation 2024/25 waren in etwa nur halb so hoch wie während des Peaks der vorhergehenden Saison 2023/24, während die B/Victoria Viren die bisher stärkste Welle hatten. Dieses Zirkulationsmuster lässt sich aus der Immunitätslage der vorhergehenden Saisons erklären, die der Pandemie folgten. 2024/25 war die dritte Grippewelle nach der COVID-19 Pandemie und die erste dieser drei Wellen, in welcher sich das Zirkulationsmuster der sonst für Influenzaviren üblichen Aktivität mit stärkerer Zirkulation Mitte Januar bis Ende März angepasst hatte.
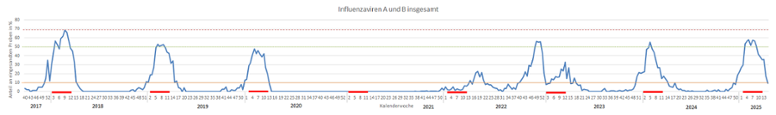
Abb. 13: Kumulative Nachweisraten von Influenza A und B Viren 2017/18 bis 2024/25; rote gestrichelte Linie: Peak der Viruszirkulation 2024/25 zum Vergleich der Peaks der Nachweisraten; grüne gepunktete Linie: Nachweisraten von 50% zum Vergleich der Dauer stärkerer Influenzavirusaktivität; ockerfarbene Linie: Nachweisraten von 10%; roter Balken: Zeit von Mitte Januar bis Ende März
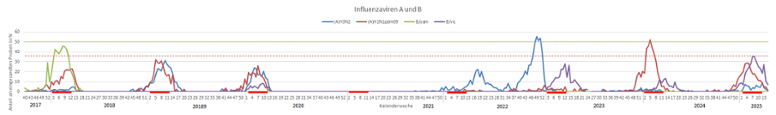
Abb. 14: Nachweise von Influenza A und B Virus Subtypen und Linien 2017/18 bis 2024/25; rote gestrichelte Linie: Peak der B-Viruszirkulation 2024/25 zum Vergleich der Peaks der Nachweisraten; grüne gepunktete Linie: Nachweisraten von 50% zum Vergleich der Dauer stärkerer Influenzavirusaktivität%; roter Balken: Zeit von Mitte Januar bis Ende März
